細菌と真菌が混在したマイクロバイオームの定量解析を可能に
記事配信日:
2025/03/18 14:00 提供元:共同通信PRワイヤー
ポイント
・ 次世代シーケンサーによるマイクロバイオーム解析のための12種類の人工核酸標準物質を開発
・ 標準物質を試料に添加することで、細菌など原核生物と真菌など真核微生物の比較、絶対定量が可能
・ これまで実現できていなかった真菌などを含めたより詳細なマイクロバイオーム研究に貢献
【画像:https://kyodonewsprwire.jp/img/202503145772-O1-B7uk965n】
概 要
国立研究開発法人 産業技術総合研究所(以下「産総研」という)バイオメディカル研究部門 Tourlousse Dieter主任研究員と 関口勇地 総括研究主幹は、細菌や真菌が混在した多種類の微生物種で構成されるマイクロバイオーム(複合微生物相)の定量解析を可能とする人工核酸標準物質を開発しました。
自然環境やヒトの腸内などで見られるマイクロバイオームの大部分は、細菌等に加えて真菌などの真核微生物を含むさまざまな種類の微生物種から構成されています。マイクロバイオームの解析において、それを構成する微生物種を同定し、その存在量を明らかにすることは重要です。しかし、次世代シーケンサーを用いた解析の結果からは微生物種の同定とその存在比が得られるものの、その絶対量を直接測定することはできません。これは、異なる試料間や研究間でマイクロバイオーム解析結果を比較、検討する上で大きな課題でした。
産総研ではこれまでに、次世代シーケンサーを用いて、細菌など原核生物の絶対量の定量を可能とする人工核酸標準物質を開発してきました。今回、真菌などを捕捉可能な人工核酸標準物質を開発し、それらをこれまでに開発した人工核酸標準物質と組み合わせて利用することで、その対象範囲を真菌などの真核微生物まで拡張することに成功しました。
真菌やその他の真核微生物は、腸内の微生物の1%未満に過ぎませんが、クローン病などの疾患の重要な要因として認識されつつあります。本技術により、マイクロバイオーム中の細菌と真菌の絶対量を同時測定することが可能となります。こうして得られる絶対量は、疾患などに関わるマイクロバイオーム情報と健康者のそれとを比較する際に有用であり、新しい診断薬や治療薬の開発への活用が期待されます。
なお、この研究成果の詳細は2025年3月17日に「ISME Communications」に掲載されました。
下線部は【用語解説】参照
開発の社会的背景
マイクロバイオームは、土壌や河川など特定の環境やヒトをはじめ生物の体内に存在する微生物群集と、その遺伝情報の集まりです。近年特に、ヒトの腸内におけるマイクロバイオームの多様性や安定性と、糖尿病などの代謝性疾患や自閉症などの神経疾患、アレルギーや免疫疾患との関連が指摘され、研究が進んでいます。また、ヒトの腸内に存在するマイクロバイオームには細菌だけでなく酵母などの真菌も存在し、これらは複雑に相互作用しています。真菌やその他の真核微⽣物は腸内の微⽣物の1%未満に過ぎませんが、クローン病などの炎症性疾患のリスクを高める場合がある一方で、その代謝物が細菌の成長を支える要素にもなっています。また、真菌はヒトの皮膚常在微生物としても重要です。それら真菌の役割を詳細に理解することで、腸内マイクロバイオームを標的とした新しい治療法や予防法の開発につながる可能性があります。
こうした研究を推進するためのマイクロバイオームの解析には、多くの場合次世代シーケンサーが用いられます。その解析結果からは試料中の微生物種の同定とその存在比が得られるものの、その絶対量を直接測定することはできません。そのため、これまで原核生物と真核微生物の存在量を直接比較することは不可能でした。これは異なる試料間や他者の研究結果と比較、検討する上で重要な問題となります。
研究の経緯
産総研は、次世代シーケンサーを用いたマイクロバイオーム解析の信頼性向上のため、解析結果の精度管理に資する研究開発を行っています。これまで、マイクロバイオーム試料に適用できる精度管理用の標準物質の開発を行ってきました(2016年12月14日 産総研プレス発表)。今回、この技術と組み合わせて細菌だけでなく真菌も同時に絶対定量できる標準物質を開発しました。
研究の内容
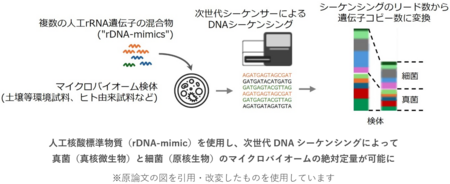
本研究では、次世代シーケンシング技術を利用したリボソームRNA(rRNA)遺伝子解析を通じ、真核微生物および原核生物のマイクロバイオームを同時に解析、その結果を比較、また絶対定量を行うための人工核酸標準物質を開発しました。rRNA遺伝子はウイルスなどを除くすべての生物が保有しており、真菌および細菌のマイクロバイオームの研究において広く用いられているマーカー遺伝子です。ただし、真核微生物と原核生物ではその遺伝子の構造や塩基配列がお互いに大きく異なり、両者を同時にPCRで増幅できるユニバーサルPCRプライマーは存在しません。そのため、真核微生物、原核生物のマイクロバイオームは、異なるPCRプライマーで増幅し、その塩基配列を次世代シーケンサーで解析し、真核微生物の微生物構成の相対定量値、原核生物の微生物構成の相対定量値をそれぞれ別々に取得します。これらの情報はそれぞれ相対定量値であるため、真核微生物と原核生物の存在量を相互に比較することができず、その比率を推定するためには各分類群のrRNA遺伝子のコピー数を別に定量する必要がありました。すなわち今までの方法では、次世代シーケンサーによる真核微生物rRNA遺伝子、原核生物rRNA遺伝子の解析と共に、真核微生物由来rRNA遺伝子コピー数、原核生物由来rRNA遺伝子コピー数を別の定量PCR等で推定する必要があります。このような煩雑さを解消するため、われわれは両系統分類群の相対定量値をつなぐための人工核酸標準物質(rDNA-mimics)を作製しました。
rRNA遺伝子はいくつかの領域から構成されていて、一部は種を超えて類似性の高い領域(保存領域)ですが、生物種に応じて配列が異なる領域(可変領域)も存在します。マイクロバイオーム解析では、サンプル中のrRNA遺伝子を、真菌と細菌に分け、それぞれの保存領域に結合するPCRプライマーを用いて増幅し、その間に存在する可変領域の塩基配列に基づき生物種を網羅的に定量します。本研究で開発したrDNA-mimicsは天然に存在するいくつかのrRNA遺伝子の保存領域と同じ塩基配列を有していますが、可変領域は天然には存在しない特徴的な人工配列に置き換えています(図1)。この設計により、保存領域に結合するPCRプライマーによってrDNA-mimicsをPCRで増幅することが可能となる一方、可変領域が自然界に存在しない塩基配列になっているため、バイオインフォマティクスによる解析時に他の生物由来rRNA遺伝子配列と容易に識別することが可能です。本研究で設計・合成した計12種類のrDNA-mimicsは、いずれも真核微生物のrRNA遺伝子による次世代シーケンシング解析で広く利用されるPCRプライマーと結合する保存領域を有しています。また、このうちの2種類は、真核微生物解析用の保存領域および人工配列と共に、原核生物のrRNA遺伝子を解析するための保存領域および人工配列を有しています。これらのDNA混合物を解析対象のマイクロバイオーム試料に添加すると、その定量値を基準として、真核微生物と原核生物の次世代シーケンシング解析による相対定量値から両者の比率を推定でき、存在量を比較するための追加の定量PCRが不要になります。
【画像:https://kyodonewsprwire.jp/img/202503145772-O2-dM4Qh8Jq】
実際、rDNA-mimicsの混合物をDNA抽出の際にマイクロバイオームサンプルに直接添加したところ、シーケンシング後、rDNA-mimicsと検体由来rRNA遺伝子の両者の配列と、その存在量が決定されました。さらに、検体に添加されたrDNA-mimicsの物質量(あるいはコピー数)から検出された微生物由来rRNA遺伝子数を、絶対量に変換することもできました(図2右上下)。このアプローチにより、従来の方法で得られる相対存在量データだけでは達成できなかった、2つの大きな分類群(真核微生物、原核生物)にまたがるマイクロバイオーム解析がrDNA-mimicsにより可能になることを実証しました。
【画像:https://kyodonewsprwire.jp/img/202503145772-O3-E86M8hpT】
さらに、rDNA-mimicsはマイクロバイオーム解析の信頼性を評価できることも実証しました。rDNA-mimicsは、その添加濃度に応じて次世代シーケンシングで検出される存在量が正しく比例することが確認されました(図2中央下グラフ)。このことは、例えば、複数のrDNA-mimicsを異なる濃度で混合したrDNA-mimics混合物を作製して毎回の次世代シーケンシング解析に添加、予想される存在量比とシーケンシングで測定される存在量比を比較することで、毎回の解析の定量性に関する精度を評価することができる可能性を示しています。
今後の予定
私たちは、本手法による絶対定量と解析の信頼性を保証することを活かして、ヒトマイクロバイオームに関連するさまざまな研究開発を進めるとともに、新たなマイクロバイオーム解析技術の開発を進めています。今後は、rDNA-mimicsを用いて人の皮膚に存在するさまざまな種類の真菌や細菌の絶対定量を行い、品質の高いデータベースの構築を進めます。さらに、人工核酸標準物質などを用いてマイクロバイオーム解析の精度管理技術を向上させ、マイクロバイオーム解析の質保証、標準化を進める予定です。また、ショットガンメタゲノム解析を含むさまざまなマイクロバイオーム解析技術を想定して、人工核酸標準物質の適用範囲の拡大を行う予定です。
論文情報
掲載誌:ISME Communications
論文タイトル:Synthetic DNA Spike-in Standards for Cross-Domain Absolute Quantification of Microbiomes by rRNA Gene Amplicon Sequencing
著者:Dieter M. Tourlousse & Yuji Sekiguchi
DOI:https://doi.org/10.1093/ismeco/ycaf028
用語解説
マイクロバイオーム(複合微生物相)
特定の環境中に存在する微生物群や、そのゲノム、あるいはそれらの持つ遺伝子の集合体を指す。具体的には、人間の腸内マイクロバイオームや土壌マイクロバイオーム、海洋中のマイクロバイオームなどがある。
次世代シーケンサー
従来のシーケンサーとは異なり、一度に読み取れる塩基配列の長さが50~500塩基(従来法では約800塩基)と短いものの、高度並列処理により1回の解析で数千万から数十億塩基対の塩基配列情報を得ることができるシーケンサー。
絶対定量
rRNA遺伝子などの分子の存在量を単位試料重量あたりの分子数など、絶対量として定量すること。
シーケンシング
次世代シーケンサーで遺伝子の塩基配列の解読を行うこと。
PCR
ポリメラーゼ連鎖反応(polymerase chain reaction)。DNA分子を増幅するための技術であり、その反応により同じ塩基配列を持つ核酸分子を大量に合成することが可能。
プライマー
PCR法で利用するもので、DNAポリメラーゼが DNA を合成する際の核酸の断片。この断片に結合するDNA部位の周辺のDNA断片がPCRで増幅される。
ショットガンメタゲノム解析
rRNA遺伝子などの特定の遺伝子配列の超並列解析ではなく、試料中に存在するゲノム全体を対象に解析を行う次世代DNAシーケンサーを利用したマイクロバイオーム解析方法。
プレスリリースURL
https://www.aist.go.jp/aist_j/press_release/pr2025/pr20250318/pr20250318.html
配信会社から提供を受けたコンテンツやプレスリリースを原文のまま掲載しており、J-CASTトレンドが制作した記事ではありません。お問い合わせは配信会社・プレスリリースの配信元にお願いいたします。
